Un'esclusiva per i lettori di Apitalia Online
[Condizioni di accesso ai contenuti di Apitalia Online]
| [Indice degli speciali online] |
| Scrivi il tuo commento a questo articolo! |
1 commenti disponibili  [Leggi commenti] [Scrivi commento] [Leggi commenti] [Scrivi commento] |
| Ricerca | ||||||||||||||||||||||||||
| Biodiversità di Apis mellifera in Europa | ||||||||||||||||||||||||||
| di Raffaele Dall’Olio e Marco Lodesani | ||||||||||||||||||||||||||
| Le api sono una componente vitale degli ecosistemi e sono fondamentali per il loro funzionamento. Perdere parte della loro biodiversità significherebbe mettere a rischio la sopravvivenza di un gran numero di specie botaniche che dipendono dalle api per il loro successo riproduttivo. Dal presente studio nasce un database dell’ape ligustica e di altre sottospecie europee disponibile presso l’INA | ||||||||||||||||||||||||||
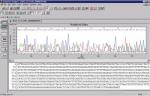
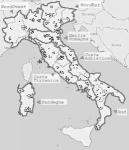
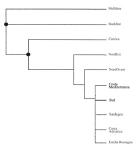
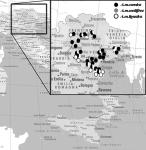
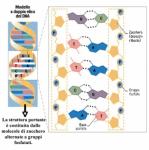 1. INTRODUZIONE 1. INTRODUZIONE1.1 La biodiversità Il termine biodiversità unisce in una sola parola il concetto di “diversità biologica” intesa come la totalità delle forme di vita presenti sulla terra. L’Articolo 2 della Convenzione di Rio de Janeiro del 1992 la definisce come “la variabilità presente negli organismi provenienti da tutti i sistemi biologici e i complessi ecologici di cui sono parte; ciò include la diversità all’interno di una specie, tra le specie e tra gli ecosistemi”. Siccome ciò che noi osserviamo è il risultato dell’interazione tra una componente genetica e l’azione dell’ambiente, la salvaguardia della biodiversità passa anche attraverso la valutazione e la conservazione della variabilità genetica esistente. In generale, la perdita di biodiversità delle specie viventi prosegue a ritmi allarmanti sia nell’UE sia a livello globale, con conseguenze potenzialmente molto gravi per la sopravvivenza e la crescita economica sostenibile. Benché la ricerca sulla conservazione della biodiversità sia una priorità europea, molti degli sforzi in questo senso sembrano poco coordinati e frammentati e questo aspetto complica la formulazione puntuale di risposte a problematiche specifiche. Nel 2003, 51 paesi europei hanno adottato la risoluzione di Kiev con l’obiettivo comune di porre termine alla perdita di biodiversità entro il 2010. La strategia per la conservazione della biodiversità della Commissione europea si propone di raggiungere l’obiettivo mediante un insieme di azioni politiche. Tuttavia, tali risposte sono gravemente compromesse dalla mancanza di una scienza efficace riguardante sia la valutazione dello status della biodiversità, sia il cambiamento e le sue implicazioni per scopi sostenibili. 1.2 Apis mellifera L. in Europa: un patrimonio da tutelare Nonostante le api siano spesso considerate animali “addomesticati”, in realtà l’uomo è riuscito solo a trasferirne l’allevamento all’interno di arnie, ma è ben lontano dall’avere addomesticato questo insetto così come ha fatto con il pollame, i maiali e altro bestiame (basti pensare che le api possono lasciare il loro alveare in qualsiasi momento per continuare a riprodursi con successo in natura). In Europa così come in tutto il resto del mondo, le api sono una parte vitale degli ecosistemi e sono essenziali per il loro funzionamento; perdere parte della biodiversità del principale insetto pronubo del pianeta, significherebbe minacciare una moltitudine di specie botaniche che affidano quasi esclusivamente alle api il loro successo riproduttivo. Tuttavia, sebbene l’importanza di Apis mellifera sia da molti riconosciuta, in pochi si rendono conto che la sua biodiversità è in pericolo, e che dunque è necessario tutelarla. Le api sono definite generaliste perché, contrariamente alla maggior parte degli apoidei solitari, bottinano su una vasta gamma di specie vegetali. Ci si potrebbe chiedere perché una specie generalista, originaria di un territorio ampio comprendente Africa, Europa e Medio Oriente, successivamente introdotta in Asia, America e Australia ed oggi diffusa in modo pressoché cosmopolita, abbia bisogno di essere tutelata. L’uomo ha introdotto e diffuso razze non autoctone di api in tutto il mondo, andando ad interferire con i naturali processi adattativi-evolutivi. Diretta conseguenza (spesso sottovalutata) di questo comportamento è una sorta di “globalizzazione genetica”, ossia la perdita di biodiversità derivante dal flusso di geni tra le colonie allevate e una popolazione selvatica sempre più esigua. L’allevamento di api nel XX° secolo - dominato dall’introduzione di sottospecie considerate superiori - ha portato all’estinzione di ecotipi locali in molte parti d’Europa. Ad esempio, in Germania la specie indigena A. m. mellifera è stata quasi completamente sostituita in seguito ad una massiccia campagna di introduzione di api regina di A. m. carnica appartenenti al ceppo austriaco-balcanico (Ruttner, 1988). 1.3 La situazione italiana Anche per l’ape italiana (A. m. ligustica), la maggior attenzione dedicata alla selezione e alla riproduzione a fini commerciali, ha progressivamente condotto a trascurare le popolazioni locali, forse economicamente meno pregiate ma portatrici di caratteristiche utili all’adattamento nei rispettivi ambienti di origine, con l’inevitabile conseguenza di metterne a rischio la biodiversità. Un caso estremo è quello della Sicilia: negli ultimi decenni la massiccia importazione nell’isola di api di razza ligustica, più adatte a un’apicoltura di tipo intensivo, ha compromesso l’integrità genetica delle popolazioni di A.m. siciliana, tanto che oggi sono in atto iniziative per il recupero e la salvaguardia dell’ape sicula in purezza. In effetti allo stato attuale delle cose, nella gran parte della Sicilia prevalgono popolazioni di api ibride tra le due sottospecie (Biondo et al., 1991; Sinacori et al., 1998). Le popolazioni di api selvatiche (o naturali che dir si voglia) in Europa e, in misura minore, in Africa, negli ultimi anni sono state fortemente influenzate dalle attività dell’uomo. Le popolazioni autoctone di api sono naturalmente soggette ad un continuo flusso genico dovuto alla loro peculiare biologia riproduttiva che - unico caso tra gli animali allevati - non consente il completo controllo dell’accoppiamento. L’area di provenienza dei fuchi, futuri padri delle operaie formanti la colonia, è infatti variabile da pochi decine ad alcune centinaia di km2. Le popolazioni selvatiche inoltre, in competizione per le risorse alimentari con quelle allevate, devono contemporaneamente affrontare in modo autonomo le malattie introdotte dalle attività dell’uomo: la loro variabilità genetica è dunque ulteriormente minacciata di depauperamento da rigidi “colli di bottiglia” dovuti proprio alle patologie. In Europa, sono quindi due le principali componenti della biodiversità di Apis mellifera minacciate: 1. la sopravvivenza di ecotipi locali; 2. la quantità totale di diversità genetica. Entrambe sono importanti sia in quanto parte dell’eredità biologica europea, sia come fonte potenziale per il futuro miglioramento dell’ape a fini zootecnici (ad esempio, la resistenza alle malattie). In questo articolo vengono discussi i risultati della ricerca svolta dall’Istituto Nazionale di Apicoltura nell’ambito del progetto BABE (“Beekeeping and Apis Biodiversity in Europe”, EVK-2000-00628): in particolare si è indagato sulla attuale variabilità genetica di A. m. ligustica sul territorio nazionale, ed i risultati sono stati organizzati in un Data Base, nella speranza che sia rappresentativo e che possa rivelarsi un importante strumento di caratterizzazione e di tutela della sottospecie (ad esempio per identificare l’introgressione, ossia l’introduzione di geni di una specie nel corredo genetico di altre specie, di tratti genomici non autoctoni e per una futura valutazione degli effetti dei programmi di allevamento). Si è inoltre cercato di correlare i dati ottenuti a potenziali fonti di alterazione del patrimonio genetico, dovute ai metodi di allevamento intensivo da passati eventi epidemici (Lodesani et al., 1995). 2. LE BIOTECNOLOGIE E LA VARIABILITA' DEL DNA: CENNI DI APPLICAZIONI PRATICHE Il genoma, è il complesso dei geni che costituiscono l’individuo, ed è un immenso “documento” che viene trasmesso da ogni cellula madre alla cellula figlia. Si pensi che il genoma nucleare umano (ossia il materiale genetico contenuto nel nucleo di ciascuna cellula somatica) è composto da circa 100.000 geni recanti serie di istruzioni per la sintesi di decine di migliaia di proteine ed è organizzato in 23 coppie di cromosomi, dove i geni sono localizzati in modo contiguo o disperso. Ogni proteina, a sua volta, è composta da centinaia di amminoacidi, ed ogni specifico amminoacido viene definito da una tripletta di basi di DNA. La sequenza di DNA quindi, attraverso un alfabeto di sole quattro lettere: A, T, C, G (corrispondenti alle basi azotate adenina, timina, citosina, guanina) contiene le informazioni necessarie per scrivere l’intero “documento” genetico individuale (Fig. 1). Si aggiunga a tutto ciò, che il DNA che serve a codificare le proteine è solo una piccola parte del DNA totale: la maggior parte del DNA, e delle relative sequenze di basi, ha una funzione strutturale o ad oggi sconosciuta. Complessivamente, il genoma umano conta circa 3 miliardi di coppie di basi disposte in una lunghissima sequenza a doppia elica (DNA), avvolta a costituire 23 coppie di cromosomi. Nel nucleo di ogni cellula umana vi è un filamento di DNA che, se interamente “srotolato”, misurerebbe oltre un metro. E che dire delle api? Cosa sappiamo del loro genoma? Il laboratorio del CNRS di Gif-sur-Yvette (Francia) è da oltre 10 anni impegnato nello studio del genoma di Apis mellifera, e nel 2002 ha pubblicato la prima generazione della mappa genetica dell’ape (Solignac M., et al. 2004). Oggi, è noto anche il genoma completo di Apis mellifera (completato presso il Baylor College of Medicine di Houston) che ha una dimensione di circa 300 milioni di coppie di basi raccolte su 16 coppie di cromosomi (Fig. 2); anche avvalendosi dei risultati del “Progetto Genoma di Apis mellifera”, è stato possibile presentare al recente congresso “Eurbee”, tenutosi a Praga nel settembre 2006 la terza generazione della mappa genetica dell’ape. I marcatori noti sono 2008 loci microsatellite, e da questi si calcola che la mappa del genoma dell’ape abbia una lunghezza di 4114.5cM, un valore molto più elevato rispetto ad altri insetti ed anche più elevato del valore noto per l’uomo (nonostante il genoma dell’ape sia 10 volte più piccolo). I marcatori oggi disponibili sono quindi estremamente densi e ben distribuiti (circa 93Kb tra un marcatore e l’altro, corrispondenti a circa 5 o 6 geni). Oltre al genoma nucleare, vi è un’altra porzione di materiale genetico che si trova al di fuori del nucleo cellulare chiamata genoma mitocondriale: il DNA dei mitocondri (organelli addetti alla respirazione cellulare presenti nel citoplasma) è una molecola di forma circolare che in Apis mellifera è costituita da 16.000 nucleotidi, tra cis ui vi sono 37 geni (Fig. 3) (Crozier e Crozier, 1993). Siccome al momento della fecondazione dei gameti, il materiale citoplasmatico che viene ereditato è esclusivamente quello della cellula uovo, la trasmissione del DNA mitocondriale avviene in forma non ricombinante per sola via materna. Le biotecnologie sono strumenti importanti per lo studio delle dinamiche e dei fattori evolutivi che agiscono sulle popolazioni naturali. Le variazioni nelle sequenze di DNA sono alle base di tutti gli studi nel campo dell’evoluzione, della biologia conservazionistica e della genetica di popolazione. Con le tecnologie molecolari si possono analizzare individui e popolazioni per determinare variazioni genetiche, flussi genici, dimensioni, parentele, fitness di popolazioni ed altro ancora. Proporzionalmente al numero di marcatori disponibili, cresce il numero di studi che ne fa utilizzo per vari fini. In particolare, un marcatore è una zona fisica identificabile di un cromosoma, la cui ereditarietà può essere controllata. Con più marcatori conosciamo, con più è possibile avvicinarsi a specifiche sequenze bersaglio nell’intero genoma. Il DNA nucleare, diversamente da quello mitocondriale ereditato esclusivamente attraverso il citoplasma della cellula uovo, viene ereditato da entrambi i genitori: nel sistema genetico aplo-diploide di Apis mellifera si assiste ad una parentela asimmetrica, dove i fuchi (aploidi) sono portatori solo di metà corredo genetico derivato da un uovo non fecondato. Marcatori molecolari di entrambi i tipi si sono rilevati utili per studiare fenomeni di introgressione genetica, il più importante dei quali riguarda l’africanizzazione delle razze europee importate in Sud America. Per le conoscenze oggi disponibili, una specifica tipologia di marcatori si è dimostrata particolarmente adatta a studi di genetica di popolazione o alla caratterizzazione di unità tassonomiche (ossia un raggruppamento di individui nell’ambito della classificazione) molto vicine tra loro quali sottospecie o razze geografiche (in questo lavoro i due termini verranno utilizzati come sinonimi). Questi marcatori sono noti come microsatelliti ((Fig. 4) e sono abbondantemente dispersi nel DNA nucleare; sono stati individuati in tutti gli organismi eucarioti in cui sono stati cercati e la loro funzione è ad oggi incognita in quanto si tratta di DNA che non viene poi tradotto in alcuna proteina (vi sono ipotesi che sostengono che abbiano un ruolo unicamente strutturale): ciò che li caratterizza è la loro elevata variabilità, non tanto in termini di composizione quanto in termini di lunghezza. Un microsatellite è infatti costituito da un breve tratto di DNA da 1 a 5 basi azotate, ripetuto in tandem per un numero imprecisato di volte, fino anche a raggiungere dimensioni di 300-400 basi azotate: questa struttura altamente ripetitiva crea difficoltà al funzionamento dell’enzima DNA-polimerasi (deputato alla replicazione del DNA) e questo determina variazioni di lunghezza molto più frequenti che in altri tratti del genoma (Fig. 5). 3. CARATTERIZZAZIONE DI APIS MELLIFERA L. NELLA STORIA La specie rappresenta il primo livello di classificazione degli individui ed è la sola categoria tassonomica che si possa considerare presente in natura e non solo una convenzione dell’uomo. Per specie si intende un gruppo di organismi simili, in grado di incrociarsi tra loro e di produrre prole fertile. Due o più specie che possiedono caratteristiche comuni vengono raggruppate in uno stesso gruppo di organismi che prende il nome di genere. Nel genere Apis, l’ape domestica - Apis mellifera L. - è la specie maggiormente oggetto di studi volti a determinarne l’origine filogenetica e le attuali relazioni tassonomiche (Fig. 6); di questa specie sono state individuate almeno 24 sottospecie suddivise in quattro grandi rami filogenetici (indicati come M, C, A e O) sulla base dell’analisi multivariata dei parametri morfologici (Ruttner, 1988), in parte avvalorati dai dati biochimico-molecolari (Fig. 7). L’ipotesi di Ruttner è supportata da numerosi studi morfometrici (Cornuet e Fresnaye, 1988, Cornuet et al., 1989, Lebdigrissa et al., 1991), mentre lavori basati sull’analisi del DNA mitocondriale (Garnery et al., 1992; Franck et al., 1998) e nucleare (Estoup et al., 1995) portarono ad una conferma parziale di tale ipotesi, in particolare in merito ai gruppi evolutivi ‘A’ (sottospecie Africane), ‘C’ (sottospecie del Nord del Mediterraneo, inclusa, Apis mellifera ligustica) e ‘M’ (sottospecie dell’Europa occidentale). Recentemente sono stati ridefiniti anche un quarto gruppo ‘O’ comprendente le sottospecie mediorientali (Franck et al., 2000), ed un quinto gruppo ‘Y’ per la sottospecie etiope A. m. yemenitica (Franck et al. 2001). È da sottolineare che per la sua posizione geografica, l’Italia ha sempre ricoperto un ruolo di rilievo nel definire la distribuzione di numerose specie (Hewitt, 1996; Taberlet et al., 1998; Hewitt, 1999), soprattutto durante le ere glaciali. Anche le api hanno sfruttato la favorevole conformazione dell’Italia per rifugiarvisi durante l’ultima glaciazione avvenuta nel Pleistocene: recenti studi sul DNA mitocondriale e nucleare, hanno infatti portato alla luce le prove di un passato contatto tra sottospecie appartenenti a differenti gruppi evolutivi, supportando quindi l’ipotesi di un’origine ibrida per l’ape italiana (Franck et al., 2000; Marino et al., 2002). In Italia esistono oggi regioni dove la sottospecie ligustica si sovrappone naturalmente con altre (A. mellifera mellifera, A. mellifera carnica e A. mellifera sicula), come dimostrato da studi sull’analisi della variabilità allozimatica (Badino et al., 1982, 1983; Manino e Marletto, 1984; Marletto et al., 1984, Comparini e Biasiolo, 1991; Nazzi, 1992) e di altri marcatori molecolari (Franck, 2004). La diffusione delle sottospecie di api è tuttavia fortemente influenzata dall’attività dell’uomo che, per l’importanza economica che questi insetti ricoprono, cerca continuamente, attraverso l’uso di nuovi ceppi, di apportare immediati vantaggi all’allevamento sul piano produttivo. La caratterizzazione genetica dell’ape ligustica rispetto alle razze confinanti assume quindi importanza prioritaria sul piano della ricerca scientifica (per giungere ad una univoca definizione e per migliorare le conoscenze sulla genetica di popolazione delle diverse unità tassonomiche) ma ha anche inevitabili ricadute sul piano applicativo: in relazione alle specifiche caratteristiche etologiche e produttive delle diverse sottospecie, la rapida determinazione della razza di appartenenza di una popolazione è di fondamentale importanza per poter iniziare corretti e mirati programmi di selezione. Infatti, soprattutto grazie ai progressi nelle tecniche di biologia molecolare, è oggi possibile investigare e caratterizzare molti aspetti della variabilità genetica presente nelle popolazioni di api, in modo da costituire una base informativa per lo sviluppo di programmi di conservazione e miglioramento della razza e degli ecotipi locali. L’apicoltura italiana ed in particolare l’allevamento delle api regine, si è sviluppata mantenendo come costante l’utilizzo e la selezione di popolazioni autoctone di ligustica, in quanto apprezzata in tutto il mondo per le sue qualità di prolificità, docilità e scarsa propensione alla sciamatura. Per queste caratteristiche e per la capacità di adattarsi ad un ampia gamma di condizioni climatiche, molti apicoltori si sono quindi dedicati con successo all’allevamento professionale di api regine di razza ligustica, esportandole in tutto il mondo da ormai oltre un secolo (Bar-Cohen et al., 1978; Woodward, 1993). Nonostante questo comprovato successo, i Posti di Ispezione Frontaliera del nostro ministero della Salute registrano, nell’ultimo decennio, una crescente importazione di regine anche da oltre oceano (Fig. 8); questo dato è imputabile sia alla fervida curiosità degli apicoltori (da sempre stimolati dall’idea di lavorare con ceppi di api dalle caratteristiche esotiche) che dalla possibilità di ottenere regine all’inizio della primavera, importandole dall’emisfero australe. Al momento non è chiaro come queste importazioni incidano sulla composizione delle popolazioni autoctone, ma, dato il successo riscontrato dall’esportazione di api di razza ligustica in tutto il mondo, l’importanza di preservarne il patrimonio genetico si giustificherebbe con le sole ragioni economiche. Inoltre, ragionando in una prospettiva più ampia, orientata alla generale salvaguardia della biodiversità, tutti gli sforzi tesi alla conservazione delle razze endemiche europee andrebbero sostenuti. 4. CAMPIONAMENTO E ANALISI Nel biennio 2001-2002, sono stati raccolti - grazie alla collaborazione di apicoltori professionisti ed amatoriali - campioni di A. m. ligustica provenienti da tutte le regioni italiane; colleghi ricercatori europei hanno contribuito a fornire i campioni di altre razze. I campioni italiani sono stati raggruppati sulla base di sette differenti macroaree geografiche (Nord-Ovest, Nord-Est, Emilia Romagna, Costa Adriatica, Costa Tirrenica, Sud e Sardegna), come in Figura 9, e tre ulteriori gruppi sono stati costituiti per includere rispettivamente i campioni di A. m. mellifera (Mellifera), A. m. carnica (Carnica) e di Buckfast, fino ad ottenere un totale di 10 “gruppi” (popolazioni) e 379 colonie: il dettaglio delle singole località di campionamento è consultabile all’indirizzo internet: www.inapicoltura.org/online/apitalia2006/sample.htm. Nel testo, nomi non in corsivo con l’iniziale maiuscola (es: ‘Carnica’), fanno riferimento a questi raggruppamenti. Tra i numerosi marcatori microsatellite disponibili in letteratura, già descritti come polimorfici (Solignac et al., 2003), ne sono stati selezionati otto per le analisi da effettuare nel presente studio: A113, A7, A88, AB24, A107, Ap43, A28 e A14. Per valutare la distanza genetica fra le popolazioni considerate, è stato utilizzato il parametro Fst che può essere applicato a qualsiasi tipo di polimorfismo genetico. Tale parametro misura la mancanza di eterozigoti nel confronto tra due popolazioni, offrendo un modo per rappresentare la struttura di una popolazione e la sua suddivisione in sottopopolazioni. Allo stesso modo viene anche utilizzato come indice indiretto della distanza genetica tra popolazioni. Più il suo valore si avvicina a zero, più le popolazioni raffrontate sono prive di struttura e cioè non possono essere considerate diverse tra loro. A titolo esemplificativo, si riporta di seguito un modo per calcolare il parametro: Fst = Vp / p (1-p) dove: Vp: varianza della frequenza allelica (p) in un insieme di n popolazioni p: frequenza allelica media di n popolazioni. I valori di Fst dipendono dal livello di raggruppamento delle popolazioni e assumono valori fra 0 (le popolazioni sono identiche) e 1 (le popolazioni sono totalmente diverse con eccesso di omozigosi nelle sottopopolazioni rispetto alla popolazione complessiva). 5. RISULTATI II loci microsatellite confermano la loro capacità di discriminare entità tassonomiche molto vicine tra loro. Nella Figura 10 si riportano i valori di distanza genetica stimata in base al parametro Fst. In base a tale parametro,il gruppo Mellifera (unico ad includere api appartenenti al ramo evolutivo ‘M’) risulta chiaramente distinto dalle altre razze in esame nel presente studio, nonostante la vasta area di campionamento (dal nord Irlanda al Sud della Francia) e la ridotta dimensione del gruppo stesso. E’ possibile fare discriminazioni anche tra razze appartenenti allo stesso ramo evolutivo: il campione di Carnica mostra infatti valori di distanza significativamente diversi rispetto agli altri gruppi costituiti da api ligustiche, tutti ascrivibili al medesimo ramo evolutivo ‘C’: in quest’ambito è importante sottolineare che in base alla stima della distanza genetica, le maggiori affinità si registrano tra il gruppo Carnica e le api ligustiche del Nord-Est. L’analisi della popolazione di Buckfast merita un discorso a parte: i valori di distanza genetica risultano bassi e non significativi, sia rispetto l’intera popolazione Ligustica che nei confronti dei gruppi di api ligustiche del Nord Italia (Nord-Est e Nord-Ovest); dalle altre analisi statistiche effettuate emerge un quadro confuso, che non chiarisce bene se la popolazione Buckfast sia costituita da individui di diverse razze o piuttosto se siano campioni tra loro omogenei ma portatori dei caratteri di più razze. Alcuni parametri (quali il maggior numero medio di alleli e gli alti valori di eterozigosità osservata) rimarcano l’elevata diversità genetica riscontrabile in questa “razza” artificiale. Ciò rivela la sua natura intrinseca; infatti, nonostante venga spesso definita come razza a sé, la Buckfast è in realtà un incrocio tra più sottospecie creato da Padre Adam, un benedettino dell’abbazia di Buckfast (UK) che nel 1921 incominciò i suoi esperimenti incrociando l’ape nera inglese Dunklen (A. m. mellifera) con l’ape italiana (A. m. ligustica). Successivamente, nei suoi viaggi in Europa e Africa, padre Adam ebbe modo di osservare il comportamento delle diverse razze. Il suo primo incrocio si arricchì, nel corso degli anni, con geni di altre razze: la mellifera francese (1930), la cercropia (1952), la anatolica (1960), la macedonica (1982) la sahariensis e la monticola (1987). L’allevamento della Buckfast è oggi diffuso in molti paesi: Nord-America, Israele, Francia, Lussemburgo, Danimarca, Svezia, Austria, Svizzera e Germania. A causa della complessità e della numerosità degli incroci coinvolti nella sua formazione, il prodotto commerciale è quindi tutt’altro che uniforme sia dal punto di vista genetico che, quindi, del valore zootecnico. Nei confronti tra le sette popolazioni (o meglio, tra i sette gruppi) di ligustica, si osserva una generale riduzione della distanza genica e della relativa significatività: al livello di dettaglio scelto per il presente studio, non è quindi possibile distinguere popolazioni locali e la variabilità genetica riscontrata, seppur elevata, è distribuita in modo omogeneo sul territorio. Cercando di rappresentare graficamente quanto appena detto, si ottiene il dendrogramma mostrato in Figura 11. I dati sin qui ottenuti, non consentono quindi di strutturare la popolazione italiana in sottopopolazioni, in quanto la variabilità genetica riscontrata entro ogni gruppo è paragonabile a quella totale. La situazione della popolazione Sardegna è a tal proposito emblematica, poichè, nonostante l’isolamento geografico, non sono state riscontrate significative differenze rispetto alla maggior parte degli altri gruppi di ligustica. (forse a causa di un intenso e continuativo commercio di api regine dalla penisola, provenienti in gran parte da pochi produttori.). Del resto, la difficoltà di una ben definita collocazione sistematica delle popolazioni isolane, era già nota da precedenti studi di caratterizzazione di Floris et al. (1998, 2002) che ipotizzano una origine comune delle popolazioni di Corsica e Sardegna da un ceppo africano; in seguito, mentre la popolazione corsa ha subito importazioni massicce di api provenienti dalla Francia, le popolazioni sarde - soprattutto quelle meridionali per la presenza di aziende di grosse dimensioni - avrebbero fortemente risentito del flusso di importazioni dalla penisola e quindi di una continua introgressione di caratteri propri della razza ligustica. Essendo un valido metodo diagnostico per la caratterizzazione a livello sottospecifico, lo studio dei loci microsatellite trova immediata applicazione nella individuazione di popolazioni ibride, nelle indagini sulla purezza delle linee di A. m. ligustica destinate all’allevamento o nei controlli sul materiale importato. In merito a queste ricadute applicative, nel presente studio si è voluto approfondire la composizione degli ibridi artificiali Buckfast, e delle api del gruppo Nord-Est (comprendente anche campioni provenienti da zone riconosciute di ibridazione naturale); si è ritenuto dunque opportuno passare dal livello di analisi popolazionistico a quello individuale, ed ogni individuo dei gruppi Nord-Est e Buckfast è stato messo a confronto con campioni di riferimento delle tre sottospecie A. m. ligustica, A. m. carnica, e A. m. mellifera, cercando di ottenere un quadro delle caratteristiche genetiche individuali. Tutti i campioni del gruppo Buckfast riportano caratteristiche genetiche riconducibili a tutte e tre le sottospecie di riferimento, confermandone quindi l’origine marcatamente ibrida. Anche molti individui del gruppo Nord-Est hanno riportato caratteristiche non consone ai riferimenti in nostro possesso per la razza ligustica, in particolare mostrando affinità alla razza carnica (Fig. 12): il fatto non sarebbe stato così sorprendente se gli individui portatori di tali caratteri fossero stati raccolti esclusivamente in Friuli (già nota zona di ibridazione naturale tra le due razze); molti dei suddetti campioni provenivano invece dalla regione Veneto. Non emergendo tuttavia un chiaro gradiente geografico della distribuzione di questi caratteri, non è possibile chiarire se il dato riscontrato sia dovuto ad una naturale espansione della zona di ibridazione, a migrazioni spontanee o piuttosto una concomitanza di fattori tra cui anche le pratiche di apicoltura non stanziale e utilizzo di ceppi non autoctoni per l’allevamento. Alla luce di questo risultato si può però affermare che la tecnica adottata nel presente studio, oltre che ad esser utile strumento per la caratterizzazione sottospecifica, ha trovato immediate applicazioni pratiche nell’identificazione di campioni ibridi ed eventi di introgressione genica. 6. DISCUSSIONE E CONCLUSIONI 6.1 Considerazioni generali In passato, gli agricoltori basavano la loro attività sulle varietà e razze locali (animali e vegetali), cioè un numero estremamente elevato di specie che si erano sviluppate, adattandosi allo specifico ambiente produttivo. Questo tipo di selezione non spinta presentava una differenza principale rispetto alla selezione delle razze cosmopolite, ossia considera il legame esistente tra l’evoluzione animale e l’ambiente in cui vive e produce. Quindi, ognuna delle razze o popolazioni autoctone si è armonicamente integrata con il proprio ambiente, con il clima, i pascoli e la flora che li caratterizzano. Per questo motivo possiamo dire che la selezione delle razze autoctone ha prodotto animali adatti a indirizzi produttivi differenziati, armonicamente inseriti nel contesto ecologico. Il concetto di selezione è comunque importante. Tutte le razze allevate sono il frutto della selezione operata dall’uomo. Non è la selezione che di per sé è negativa. E’ l’obiettivo di selezione utilizzato dai selezionatori che spesso non concorda con i principi della conservazione della biodiversità e dell’agricoltura sostenibile producendo animali più esigenti dal punto di vista alimentare e più bisognosi di cure veterinarie, quindi animali peggio adattati all’ambiente. Quando la separazione geografica limita o impedisce la libera riproduzione tra le popolazioni, inizia un processo di differenziazione genetica che diviene via via più accentuato sino alla formazione delle razze geografiche che hanno quindi una fisionomia abbastanza definita: un gruppo di individui della stessa specie che abita un certo territorio e che è geneticamente diverso da altri gruppi confinanti, geograficamente delimitati. Quindi ogni razza geografica è il prodotto di una lunga evoluzione temporale che ha determinato un equilibrio fra i componenti del proprio patrimonio genetico (coadattamento). L’incrocio fra individui geneticamente distanti (come di solito è per quello inter-razziale es. la Buckfast) rompe il coadattamento e produce nuove combinazioni genetiche (Fig. 13); nel corso del tempo queste combinazioni potrebbero riadattarsi, creando poi novità genetiche (tipo l’ape africanizzata), oppure risultare non adatte e quindi estinguersi. Nello specifico dell’apicoltura, con la crescente minaccia di un potenziale inquinamento genico da parte di razze artificiali, di materiale importato e di sottospecie naturalmente confinanti, appare opportuno pianificare in maniera cauta e costante i programmi di allevamento, così da preservare il patrimonio genetico della sottospecie A. m. ligustica, con vantaggi sia per il settore apistico nazionale sia per il mantenimento della biodiversità naturale. Alcune pratiche dell’apicoltura professionale possono infatti fortemente interferire con la composizione genetica delle popolazioni naturali e con i tentativi di preservare le razze autoctone di api: è già stato dimostrato come i programmi italiani di allevamento controllato e coordinato, che prevedono l’allevamento intensivo di migliaia di regine prodotte da poche madri selezionate, siano stati causa di perdita di diversità genetica all’interno delle popolazioni native (Bolchi-Serini et al., 1982-3; Marletto et al., 1984). La variabilità genetica delle popolazioni di api presenti sul territorio nazionale, frutto di un lungo processo evolutivo e di un naturale adattamento alle diverse condizioni ambientali, costituisce un patrimonio biologico da tutelare e valorizzare sia nell’interesse dell’apicoltura che, più in generale, dell’agricoltura e dell’ambiente. La ricchezza di caratteristiche biologiche ed etologiche riscontrabili nelle popolazioni nell’ambito di una singola specie è espressione “tangibile” della diversità genetica: quest’ultima, oltre ad avere un’importanza fondamentale da un punto di vista ecologico, sta assumendo importanza anche nel settore zootecnico come possibile alternativa ai tradizionali criteri di miglioramento basati sulla selezione i quali, focalizzandosi su pochi caratteri di importanza economica, tendevano a condurre ad una sorta di omogenizzazione genetica delle popolazioni allevate. |
||||||||||||||||||||||||||
|
Ringraziamenti
Questo studio è stato co-finanziato nell’ambito del progetto BABE (EVK-2000-00628). Si ringraziano in modo particolare tutte le Associazioni ed i singoli apicoltori che hanno provveduto ad inviare i campioni per l’analisi genetica |
||||||||||||||||||||||||||
| IMMAGINI ALLEGATE A QUESTO ARTICOLO: 13 tot. | ||||||||||||||||||||||||||
|
||||||||||||||||||||||||||
| Scrivi il tuo commento a questo articolo! |
1 commenti disponibili  [Leggi commenti] [Scrivi commento] [Leggi commenti] [Scrivi commento] |
 © Apitalia - Tutti i diritti riservati © Apitalia - Tutti i diritti riservatiScritto in data 18/12/2006 da Raffaele Dall’Olio e Marco Lodesani C.R.A. - Istituto Nazionale di Apicoltura |
| COMMENTI A QUESTO ARTICOLO: 1 tot. [Leggi i commenti] |
|
|
| [Torna ad inizio pagina] |